CORONAVIRIDAE - LES CORONAVIRUS
d'après le cours de Luis ENJUANES, Departement
of Molecular and Cell Biology,
CNB, Campus Universidad Autonoma Cantoblanco, Madrid, SPAIN
INTRODUCTION
L'ordre des Nirovirales inclus 3 familles
virales : les Coronaviridae (Coronavirus), les Arteriviridae
et, probablement bientôt, les Ronoviridae, une variété
de virus ayant une forme de baguette (rod en anglais) infectant les
invertébrées tel que les crevettes.
Les Coronaviridae comprennent les genres Coronavirus
et Torovirus. Les points communs et les divergences de ces
2 genres sont les suivantes:
Caractéristiques |
Coronavirus |
Torovirus |
| Enveloppé |
+ |
+ |
| ARN positif simple brin avec queue polyA |
+ |
+ |
| Gène 5'Polymerase - Gènes
de protéines structurales en 3' |
+ |
+ |
| Le 3'co-terminal naissant porte au moins
4 ARNm subgénomique |
+ |
+ |
| Seule la région 5' de l'ARNm est
traductionnellement active |
+ |
+ |
| Décalage du cadre de lecture ribosomal
dans le gène de la polymérase |
+ |
+ |
| Une protéine M avec au moins 3 séquences
traversant la membrane |
+ |
+ |
| Bourgeonnement intracellulaire |
+ |
+ |
| Taille du génome en kB |
27-31,5 |
~25 |
| Séquence 5' Leader |
+ |
- |
| Core (Coeur) |
+ |
- |
| Nucléocapside |
Hélicoidale |
Tubulaire |
| Spicules (spike) proéminentes |
+ |
+ |
| Structure en hélice/hélice
des spicules |
+ |
+ |
Tableau 1: Caractéristiques des coronavirus
et des torovirus
I- PROPRIETES BIOLOGIQUES
Les Coronavirus (appelé ainsi car présentant
une forme de couronne ou corona) infectes les oiseaux et beaucoup de
mammifères, y compris les humains. La trachée respiratoire,
les organes gastro-intestinaux, ainsi que les tissus neurologiques sont
les cibles les plus fréquentes des coronavirus, mais
d'autres organes incluant le foie, le coeur, les reins et les yeux peuvent
êtres également affectés (Escor et al., 2001). Les
cellules épithéliales sont les cibles principales des
coronavirus (Alonso et al.). Les cellules largement ditribuées
telles que les macrophages sont aussi souvent infectés par les
coronavirus. Ces virus sont relativement restreint dans leur
spectre d'hôte, infectant seulement leur hôte naturel, et
des espèces animales relativement proche. Occasionnellement,
l'infection par le coronavirus passe la barrière d'espèce
, comme lors d'infection de dindon par le coronavirus bovin
(BCoV), ou l'infection expérimentale de chien par le TGEV. C'est
peut être ce qu'il s'est passé avec la SRAS (Syndrome Respiratoire
Aigüe Sévère). Les vecteurs biologiques sont inconnus.
Les transmissions par respiration, fécal et oral sont courantes.
Les infections de l'homme et des animaux par les coronavirus
semblent êtres ubiquitaires, car l'évidence de l'infection
a été obtenue dans tous les pays où des études
sérologiques et virologiques ont été effectuées.
II- MORPHOLOGIE
Les Coronavirus tire leurs noms de leur forme caractéristique
en couronne (Corona en latin):

Coronavirus (tiré du site du département
de Penn State Biology )
Les virons sont enveloppés et de forme sphérique; certain
Coronavirus font couramment 120-160nm de diamètre avec
un coeur interne , parfois icosaédrique, de 65nm, et une nucléocapside
en hélice..

Figure 1: Représentation schématique d'un coronavirus
Les coronavirus et les toronovirus ont tous
deux de larges projections à leurs surfaces formées de
glycoprotéines possédant un globule et une portion fine.
Les peplomers (trimère de spicule) ont environ 20nm de long chez
les coronavirus et légèrement plus petit chez
les torovirus. Chez certain coronavirus comme le BCoV et le
virus de l'hépatite murin (MHV), une seconde couche de peplomers
formée par des protéines Hémaglutinine Estérase
(HE) est également observée (Escors et al., 2001).
Un vide séparant le core interne de l'enveloppe a été
observé chez les Coronavirus en utilisant le microscopie
à cryoelectron. Le Core peut être isolé après
traitement. La décompositions de ces cores révèle
des nucléocapsides contenant des hélices de N-protéines.
III- LES PROTEINES VIRALES
Les virions contiennent de grande glycoprotéines
de surface (ou Spicule, S), des protéines intégrées
à la membrane (M), lesquelles ont intégrés dans
l'enveloppe virale 3 ou 4 segments, une petite protéine membranaire
(E), et une protéine de nucléocapside (N).
Protéine |
Coronavirus |
Torovirus |
| Glycoprotéine Spicule
(Spike) |
S |
180-220 |
200 |
| Protéine Membranaire (Membrane protein) |
M |
23-35 |
27 |
| Protéine de Nucléocapside
|
N |
50-60 |
19 |
| Petite protéine d'Enveloppe |
E |
9-12 |
N.C. |
| Protéine d'Hémagglutinine-Estérase
(Haemagglutinin-esterase) |
HE |
65 |
65 |
Tableau 2 : Les protéines associées au virus des
Coronaviridae (poid moléculaire en kDa)
Le ratio des protéines S:E:M:N varie selon
différentes sources. Pour le TGEV, ce ratio a été
estimé à 20:1:300:140, respectivement. La protéine
S est d'une taille importante comportant entre 1160 et 1452 acides
aminés, et chez certain coronavirus, est clivée en 2
sous-unités S1 et S2 (Gonzalez et al., 2002). La protéine
S est responsable de l'attachement au cellule, de l'hémaglutination,
de la fusion membranaire et de l'induction de la neutralisation des
anticorps. L'immunisation avec S seule peut induire la protection
contre d'autre coronavirus (MHV, TGEV). La protéine S a la
moitié de sa partie C-terminale en structure hélice/hélice
(coiled-coil). La protéine M possède entre 225 et 260
acides aminés et peut induire l'interféron-a.
Une sous-partie des coronavirus possède une protéine
d'Hémagglutinine-Estérase (HE) qui forme de petites
projections.de surface. Cette protéine apparemment non-essentielle
possède un domaine de fixation de récepteur pour l'acide
9-O-neuraminique-acétylé, une activité d'hémaglutination
et également des activités de destruction du récepteur
(neuraminate-O-acétylestérase). La protéine HE
montre une séquence identique à celle de la protéine
d'Hémagglutinine-Estérase du virus C de la grippe. La
protéine E (80 à 109 acides aminés), avec la
protéine M, joue une rôle essentiel dans l'assemblage
des particules de coronavirus. La protéine N ( d'une taille
comprise entre 377 à 455 acides aminé ) est une phosphoprotéine
hautement basique qui module la synthèse d'ARN viral, se fixe
à l'ARN viral et forme une nucléocapside en hélice.
Les protéines non-structurales sont généralement
non-essentielles pour la réplication du virus in vitro
ou in vivo. Une des protéine non-indispensable non-structurale
est la réplicase codée par le gène 1, qui constitue
les 2/3 du génome (18 à 22 kb). Le gène de la
réplicase a été prédi pour coder pour
une protéine d'environ 740 à 800 kDa laquelle subit
des maturations transitoires. Le gène rep code pour
2 cadres de lectures (ORFs), 1a et 1b, lesquels sont chevauchés
par quelques nucléotides. ORF 1b est dans le cadre de lecture
-1 . Plusieurs domaines du gène rep ont des fonctions
prédites basées sur les homologies de séquences:
des fonctions protéases à cystéine papaïne-like,
une protéase 3C-like chymotrypsine-picornavirus, une protéine
de facteur de croissance riche en cystéine, une polymérase
à ARN ARN-dépendante, un domaine hélicase/Fixation
de nucléoside triphosphate (NTP) et un domaine en doigt de
zinc.

Figure 2 : Comparaison des motifs du gène rep identifiées
chez les coronavirus et les arterivirus
Les autres protéines non-structurales varie
dans leurs noms et leur localisation pour la plus part de coronavirus.

Figure 3 : Représentation du génome du TGEV et motif
d'expression des ARNm
Les ORF sont représentées par des boites.
Les protéines codées par les ORF sont indiquées.
Les séquences 5'Leader sont représentées
par une petites boite bleue en 5' de chaque
gène. La queue Poly(A) est indiquée par des AAA.
Un ARN négatif complémentaire de chaque ARNm a été
détecté. S est la protéine de Spicule, M, la
protéine membranaire, N, la nucléocapside. 3a, 3b et
7 sont des protéines non-structurales.
Le gène N est habituellement localisé
en 3'terminal du génome des coronavirus, sauf pour le TGEV,
le coronavirus félin (FCoV) et le coronavirus canin (CCoV),
chez lesquels le gène N est suivi par 1 ou 2 autres gènes.
IV- ORGANISATION DU GENOME
Le génome est constitué d'un unique brin
linéaire d'ARN de polarité positive. L'ARN génomique
est l'in des génomes viral à ARn les plus grand avec une
taille comprise entre 27,6 et 31,5kb. L'ARN des Coronavirus
un cap en 5'Terminal suivi d'une séquence Leader de 65 à
98 nucléotides et d'une région non-traduite de 200 à
400 nucléotides (cf figure 3). En 3'terminal
du génome se trouve une séquence non-traduite de 200 à
500 nucléotides suivie d'une queue Poly(A). Les fonctions de
l'ARN du virion sont de faire les ARNm et de participer à l'infection.
L'ARN du virion contient entre 7 à 10 gènes fonctionnels,
dont 4 ou 5 codent pour les protéines structurales. Les gènes
sont arrangés dans un ordre précis 5'-Polymérase-(HE)-S-E-M-N-3',
avec un nombre variable d'autres gènes qui sont apparemment non-structuraux
(ns) et souvent non-essentiels, du moins en culture cellulaire. Cet
arrangement des gènes s'applique également pour les Torovirus
et les Artevirus.
Environ 2/3 de l'ARN viral est occupé par le gène de la
polymérase. Au chevauchement (overlap)entre les ORF (Open Reading
Frame ou Cadre de Lecture Ouvert) des régions 1a et 1b, il y
a une séquence "slippery" (glissante) spécifique
de 7 nucléotides et une structure en pseudonœud (signal
d'un décalage de lecture ribosomale), qui sont nécessaire
pour la traduction de l'ORF 1b. Le tiers restant du génome comprend
les gènes codant pour les protéines structurales et les
autres protéines non-structurales. L'organisation des gènes
des protéines non-structurales, qui sont intercalées entre
les gènes des protéines structurales, varie de manière
significative selon les différent Coronavirus. Un structure
en pseudo-nœud est également prédite en 3'-Terminal
de l'ARN des coronavirus.
Les Nidovirales ont été comparés à
d'autre virus à ARN+: la majorité des virus à ARN+
peuvent se regrouper dans 2 grands supergroupes: les Picornavirus-like
et les Alphavirus-like. Chacun de ces supergroupes est caractérisé
par une position ordonnée de 3 domaines conservés formant
la colonne vertébrale des polyprotéines réplicatives.
Pour les virus du supergroupe des Picornavirus-like, ces domaines inclus
une hélicase-ARN (superfamille 2 ou 3), une protéase à
cystéine (3Clpro), et une polymérase à ARN ARN-dépendante
(RdRp). Pour les virus du supergroupe des Alphavirus-like, ces domaines
inclus une methyltransférase, une hélicase-ARN (superfamille
1) et une RdRp. Le plan génétique des Nidovirus basiques
inclus une des régions non-traduite en 5' et une région
3' flanquée de 6 à 11 ORFs, certaines d'entre se chevauchant
partiellement. Les ORF1a et 1b, les plus proche du 5', sont les plus
importantes et se chevauchent. L'ORF1a code pour la polyprotéine
pp1a, et ORF1a+ORF1b code directement, grâce à un décalage
de lecture ribosomale, pour la polyprotéine pp1ab. Ces deux polyprotéines
sont organisées autour d'un cadre de travail (framework) d'une
douzaine de domaines réplicatifs et de traitement autocatalytiques
de nombreux produits au niveau des jonctions interdomaines. Les autres
ORFs, dont le nombre varie selon les virus, sont localisé plus
en aval et dirige la synthèse des protéines de capsides
et, optionnellemnt, des protéines accessoires. Pour exprimer
ces ORFs, une série de d'ARMsgm naissant est générée.
Les caractéristiques des différents domaines, en particuliers
ceux la réplicase des membres des Nicovirales, ne présentent
pas de grande séquences similaires avec ceux des autres groupes
de virus. Toutes ces observations sont compatible avec avec la théorie
d'une divergence d'évolution entre les corona-, les
arteri- et les ronivirales de la racine commune. C'est
pour cela que ces familles ont été unies dans l'ordre
des Nicovirales (Gorbalenya,2001).
V- LA REPLICATION DES CORONAVIRUS

Figure 4 : Modèle de la réplication des coronavirus
A - Attachement et pénétration:
Bien que les coronavirus puissent s'attacher aux cellules
grâce aux formes ubiquitaires acétylées des glycoprotéines
et des lipides, une fixation plus spécifique entre le virus et
un récepteur cellulaire est requise pour l'établissement
d'une infection virale. Les coronavirus sont divisés en 3 groupe.
Les membres du groupe 1 des coronavirus (qui comporte le TGEV et le
HCoV-229E) utilise l'aminopeptidase N (APN ou CD13) comme récepteur
pour l'entrée dans la cellule. Les coronavirus du groupe 2 (qui
comporte le MHV), utilise comme récepteurs des membres de la
sous-famille des glycoprotéines billiaires (bgp) de la famille
des antigène carcinoembrionique (CEA). Les gycoprotéines
contenant de l'acide scialique (acide N-acetyl-9-O-acetylneuraminique)
sont probablement un composant des molécules de la surface cellulaire
requises pour l'infection par BCoV et HCV-OC43. Néanmoins, la
fixation aux récepteurs bgp ou APN n'est pas suffisante pour
l'infection virale et n'explique pas les différences de tropisme
des coronavirus. En plus des récepteurs susmentionnés,
un second facteur mis en cause dans la protéine S (peut être
un second site de fixation) a été impliqué dans
le tropisme des Coronavirus.
B - Traduction primaire:
Pour tous les virus à ARN positif, le premier évènement
de synthèse macromoléculaire suivant l'entrée du
génome viral dans le cytoplasme est la traduction du génome
viral ARN pour produire l'ARN polymérase ARN-dépendante,
laquelle est traduite à partir du gène 1. Chez tous les
coronavirus, le gène 1 contient 2 ORFs chevauchantes (1a et 1b)
lesquels peuvent potentiellement être traduite en une protéine
de plus de 700 kDa (rep1ab) grâce à un mécanisme
de décalage du cadre de lecture ribosomale (ribosomal frame-shifting
mechanism).
Le produit de la traduction primaire est co et post-traductionnellement
modifié (process) en multiple protéines par des protéases
virales et cellulaires. L'inhibition de la synthèse protéique
à certains moments l'infection inhibe la synthèse d'ARN
viral., suggérant que la polymérase est probablement traduite
tout le long du cycle de réplication. L'ordre des évènement
des modifications et les fonctions des produits du gène 1 ne
sont pas encore totalement compris.
C - Transcription de l'ARN viral:
L'ARN génomique viral de brin positif est transcrit en un brin
d'ARN négatif, lequel est utilisé comme matrice pour la
synthèse des brins positifs viraux d'ARNm et de l'ARN génomique.
La synthese du brin d'ARN négatif atteint un pic plus tôt
et tombe plus rapidement que la synthèse du brin positif. Les
cellules infectées contiennent entre 10 et 100 fois plus de brins
positifs que de brins négatifs. Bien que des études initiales
suggéraient que tous les brins d'ARN négatif étaient
de la taille du génome, il est maintenant clair que les cellules
infectées contiennent aussi des des brins d'ARN négatifs
subgénomiques correspondant en longueur et en abondance relative
aux ARNm viraux. Tous les brins d'ARN négatif sont trouvé
sous les formes double brin, et aucun brin d'ARN négatifs n'ont
été trouvé sous forme libre. Le mécanisme
de synthèse et les fonctions des brins d'ARN négatifs
subgénomiques et de longueurs génomiques restent encore
sujet à controverse. Mais il est clair qu'ils servent d'amorces
pour la synthèse d'ARN subgénomiques car les intermédiaires
réplicatifs radiomarquées sont composées à
la fois des brins complémentaires de longueurs génomiques
et subgénomiques. Deux modèles de la synthèse de
l'ARN des coronavirus seront discutés plus loin.
Suivant les espèces de virus, il existe entre 5 et 7 ARNm subgénomiques
numérotés par odre décroissant. Les ARNm forment
une série emboitée avec un 3' commun. Bien que chaque
ARNm, excepté le plus petit, soient polycistroniques (contenant
2 ou plus ORF), seule l'ORF en 5'terminal est traduite (avec parfois
des exceptions).
Un trait caractéristiques de l'ARNm subgénomiques des
Coronavirus est la présence en leurs 5'terminal d'une séquence
Leader de 60 à 90 bases identique à la partie 5'terminal
de l'ARN génomique. Cette séquence Leader n'est pas présente
ailleurs dans le génome, mais entre chaque ORF du génome,
on trouve une une petite séquence coeur (short Core
Sequence ou CS), hautement conservée,
contenant une séquence de 6 et 18 nucléotides homologue
avec un séquence proche du 3' final du Leader. Cette CS est incluse
dans un domaine plus grand constituant la séquence de régulation
de la transcription (Transcription Regulatory Sequence ou TRS). Les
2 modèles favoris pour expliquer comment les ARN Leader sont
joint à chaque mRNA et comment les ARNm subgénomiques
sont générés sont illustrés ci dessous :

Figure 5 : Modèles de transcription des coronavirus
Le modèle doit etre en accord avec les observations
que, tardivement au cours du cycle réplicatif, la taille de la
cible des UV de chaque ARNm est similaire à leurs tailles physiques,
et que, dans les cellules simultanément infectées par
deux virus de souche MHV, approximativement la moitié des ARNm
ont un Leader issus d'une souche et codent les séquences de l'autre
souche.
Un des modèles pour la synthèse des ARNm de Coronavirus
postule une transcription leader-primed (amorcée par le Leader).
La transcription de l'ARN Leader commencerait en 3'terminal du brin
d'ARN négatif complémentaire et terminerait avec la dissociation
du Leader de la matrice, soit seul soit associé à une
(des) protéine(s) polymerase. Le Leader fixerait ensuite les
séquences intergéniques en aval de de la matrice négative
et servirait d'amorce (primer) pour la transcription des ARNm. Ces modèles
sont basés sur des études anciennes montrant que les cellules
infectées contiennent seulement des matrices de brin négatif
de la taille du génome. L'épissage du génome étaient
exclu car le taux d'inactivation par les ultraviolet de la synthèse
des ARN génomiques et subgénomiques était proportionnel
à la longueur de l'ARN. Ceci suggérait que l'ARN Leader
était joint au corps de l'ARNm subgénomique durant la
synthèse du brin positif. Les points obscurs de ce modèle
sont : la cause du point de terminaison de la transcription initial
du Leader et savoir si la fixation subsequent du Leader aux séquences
intergéniques est médiée par un alignement de séquence
d'ARN ou par reconnaissance par l'ARN polymérase. Plusieurs points
vont dans le sens du modèle de la transcription leader-primed
:
- Les cellules infectées par le MHV contiennent des ARN Leader
libres d'une taille de 60 à 90 nucléotides.
- Un mutant thermo-sensible du MHV accumule des ARN Leader, mais pas
d'ARNm, àtempérature non-permissive.
- Les Leader transcrits issus de 2 souches différentes de MHV
dans une même cellule sont joints au hasard aux ARNm des autres
souche.
- Dans un système de transcription in vitro, dérivé
de cellules infectées par le MHV, les ARN Leader ajouté
exogénèsement peuvent être incorporés dans
les ARNm.
Dans le modèle de transcription leader-primed,
le CS sert de promoteur pour la transcription des ARNm. Le CS est conservé
chez la pluspart des espèces de Coronavirus. Six
ou sept nucléotides à l'intérieur du CS
contitue le cœur de la séquence promotrice, et des séquences
additionnelles en amont de l'ARN Leader et en 5'terminal des ARN génomiques
sont aussi requises pour l'initiation de la transcription. Il exite
des variabilités entre différentes souches virales de
MHV sur les CS qui précèdent les même ORFs. Certain
ARNm, comme l'ARNm 2-1 du MHV, peuvent être transcrit seulement
si l'ARN Leader contient une séquence compatible. La séquence
Leader de certain ARNm diffère légèrement de la
séquence Leader de l'ARN génomique correspondant, suggérant
que le processus d'amorçage par Leader requiert des séquences
modifiées aux sites de fusion Leader-ARNm. Le modèle de
transcription leader-primed est compatible avec la génération
d'ARNm issus des matrices négatives subgénomiques et du
brin complémentaire, mais le modèle ne tient pas compte
de la génération d'observations des ARNs négatifs
subgénomiques et des ARN intermédiares de réplication
subgénomiques.
Un modèle alternatif pour la synthèse d'ARN des Coronavirus
postule une transcritpion discontinue durant la synthèse du brin
d'ARN négatif complémentaire. Durant la synthèse
du brin d'ARN négatif à partir de la matrice u génomique,
la polymérase ferait des pauses sur l'un des CS et sauterait
jusqu'au 3'terminal de la séquence Leader à coté
du 5'terminal de la matrice d'ARN génomique, générant
un ARN négatif subgénomique avec une séquence
Leader antisens en son 3'. Ces ARN négatifs subgénomiques
et les brins négatifs complémentaires servirait alors
de matrice pour une transcription ininterrompue d'ARNm et génomiques
positifs. Dans ce modèle, le CS sert de séquence de terminaison
de la transcription et/ou de séquence qui interragit avec l'ARN
Leader pour permettre les saut de la polymérase durant la synthèse
du brin d'ARN négatif. Plusieurs points observées vont
dans le sens de ce modèle:
- Les ARN subgénomiques négatifs ont une séquence
poly(U) à leurs extrémités5' et une séquence
Leader à leurs extrémités 3'. Ainsi, ils apparaissent
comme étant les copies exact et complémentaires des
ARNm viraux.
- La relative abondance des différents ARN subgénomiques
négatifs dans les cellules infectées est la même
que la relative abondance des ARNm viraux correspondant.
- Les ARN subgénomiques intermédiares de réplication
(R.I.) sont répliqués activement dans les cellules infectées.
Lorsque les ARN sugénomiques ont été séparés
par taille et dénaturés, les petit R.I. ont généré
seulement des petits ARNm, et les grand R.I. ont généré
seulement des grand ARNm. Ceci suggère que chaque ARNm subgénomique
devrait être transcrit à partir d'une matrice de brin
négative de même taille subgénomique.
Les données actuellement disponibles ne donnent pas l'avantage
à l'un ou l'autre de des modèles de transcription des
coronavirus. Néanmoin, récemment, de fortes évidences
concernant les coronavirus favoriseraient le modèle de transcription
discontinue durant la synthèse du brin d'ARN négatif (Sawicki
et al., 2001). En réalité, ces auteurs ont montré
que, en ajoutant des R.I. (Replicative Intermediate RNA) et des R.F.
natifs (Replicative Form RNA), les cellules hépatiques de souris
infectées par le virus contenaient 6 espèces d'ARN intermédiaires
(R.I.) actifs transcrivant des ARNm subgénomiques (sgmRNA). Ils
ont nommé ces intermédiares de transcrition (TIs) et les
formes transcriptives natives (TFs) car il n'y a pas de réplication
de l'ARN de taille génomique.
D - La transcription des ARNm est guidée
par l'appariement des TRSs sens et anti-sens:
Récemment, le groupe de Eric Snijder (Leiden, Hollande) a rapporté
que les arterivirus (et donc probablement les coronavirus) utilise un
mécanisme de transcription discontinue. Durant ce processus,
les ARNm générés possèdent l'ARN leader,
fusionné en des positions spécifiques en différentes
région 3' du génome. En utilisant des clones d'ADNcomplémentaires
infectieux issus du virus arterivirus equine arteritis (EAV) et mutés
spécifiquement, il a été montré que la synthèse
des sgmRNA requiert un appariement de base entre la short Core Sequence
(CS) du leader un complement du corps de la CS dans le brin complémentaire
négatif. Des mutations dans le corps (body) de la CS du RNA7
de l'EAV réduisent sévèrement voire abolie la transcription
de l'(ARN7 subgénomique. La construction de doubles mutants dans
lesquels un mutant de la CS leader a été combiné
avec le mutant correspondant du corps CS de l'ARN7 amène à
une restauration spécifique de la synthèse d'ARN7. L'analyse
des jonctions ARNm leader - body sur un grand nombre de mutants rend
favorable un mécanisme de transcription du brin négatif
discontinue ressemblant à une recombinaison copy choice-RNA (copy-choice
RNA recombination).
Le remplacement de C par G dans le Leader et le coprs (body) du gène
7 ( pour générer la CS 5'-UGAAG-3') n' a d'effet détectable
sur sa capacité de produire le sgmRNA correspondant. Ceci suggère
que la séquence de la CS n'est pas cruciale en elle-même,
tant qu'est maintenue la possibilité d'appariement par base de
la CS. Si l'on utilise des mutants dans lesquels la CS entiere (5'-UCAAC-3')
du leader a été remplacée par une séquence
complétement différentes des CS connues (5'-AGUUG-3'),
les ARNm subgénomiques ne sont plus produits.
Construction |
Leader CS |
body CS de l'ARN7 |
Production de l'ARNm7 |
Sauvage |
5'UCAAC-3' |
5'UCAAC-3' |
+ |
L1 |
UGAAC |
UCAAC |
- |
B1 |
UCAAC |
UGAAC |
- |
LB1 |
UGAAC |
UGAAC |
+ |
L2 |
UCAAG |
UCAAC |
- |
B2 |
UCAAC |
UCAAG |
- |
LB2 |
UCAAG |
UCAAG |
+ |
L3 |
UGAAG |
UCAAC |
- |
B3 |
UCAAC |
UGAAG |
- |
LB3 |
UGAAG |
UGAAG |
+ |
L4 |
AGUUC |
UCAAC |
- |
B4 |
UCAAC |
AGUUC |
- |
| LB4
a |
AGUUC
a |
AGUUC
a
|
+
(mais plus faible que le type sauvage) |
Tableau 3 : Les clones mutants des ADNc complet
d'EAV avec leurs substitutions dans le leader TRS
(van Marle et al., 1999)
La mutagénèse du corps due la CS dans certain
gène, comme le gène 3 de l'EAV, n'abroge pas la synthèse
de l'ARNm3. Ceci a été expliqué par l'identification
de 2 espèces majeurs d'ARNm3, l'une(l'ARNm3) dérivant
de la séquence consensus de la CS (5'UCAAC-3') et une autre (l'ARNm3.1)
derivant d'une sequence non-consensus (5'-UCAAUACCC-3'). Cette dernière
contient le résidu C en 5 mais cotient en plus 5 nucléotides
en aval ( UACCC-3'). Les mutants en position 5 de ce type ne sont pas
affectés par la synthèse de l'ARNm3.1, le C en position
5 n'est indispensable à l'appariement avec la sequence 3' de
la CS du Leader, les 5 nucléotides en aval semblant pouvoir remplir
ce rôle.
La CS dans les ARNsgm est dérivée exclusivement du corps
de la CS. Le mutant B1, et dans une monidre mesure les mutants L1, L2
et B2 produise encore de faible niveau d'ARNm7 (cf tableau). L'ARN7
produit par le mutant B1 a été détecté par
hybridation. On a également pu détecté de l'ARN7
produit par les mutants B2, L1 et L2 après de longues expositions
et, de manière plus convaincante, par RT-PCR. Dans ce cas là,
est ce que c'est la CS qui forme la jonction Leader-body (Leader-corps)
de l'ARNm est dérivé du Leader ou de la séquence
du corps (body). Parceque la CS du Leader et du corps (body) sont normallement
identique, cet question ne peut être résolue en utilisant
des souches sauvages. Mais des mutants des construction L1, L2, B1 et
B2 ont permis de déterminer l'origine de la sequence à
la jonction Leader-body. Celle-ci provient de la CS du corps (body CS)
acr seul les mutants B1 et B2 transmettent leurs mutations. Ceci suggère
fortement que la transcription discontinue prend place durant la synthèse
du brin négatif.
E - La synthèse discontinue
des ARN ne dépend pas seulement des appariements entre les TRS
leader sens et le corps(body) TRS anti-sens, mais également de
la séquence primaire des corps TRS:
L'ARn génomique de L'EAV contient plusieurs séquences
qui s'apparie précisement à la TRS Leader, mais qui ne
sont pas utilisées lors de la synthèse des ARNsgm. Ceci
suggère que la seule similarité entre les TRS Leader et
body soit, bien que nécessaire, non suffisante pour la synthèse
discontinue des ARN.
pour mieu comprendre ce qu'il se passe, l'équipe de Eric Snijder
a réalisé une étude par mutagénèse
dirigée du TRS du Leader et du body de l'EAV. Chaque nucléotide
de l'EAV ont été substitué avec chacune des 3 autres
nucléotides possibles.Chaque mutation a été effectué
sur la TRS du Leader, celle du Body de l'ARN7 et celle des deux TRS,
réalisant ainsi 54 mutants. Tandis que la TRS du Leader ne joue
qu'un rôle de cible dans la translocation du brin naissant, les
nucléotides de la TRS du body semble être dans des positions
très spécifiques et de nature non-interchangeable.
L'effet d'une seule mutation d'une des deux TRS (body ou Leader) était
beaucoup lié à la nature de la base, affectant la synthèse
des sgRNA7 de manière variable (cf figure).

Figure 6 : Quantité relative d'ARNsgm7 produite
par les mutants EAV de la TRS
(d'après Pasternak et al, 2001)
On remarque qu'aucun changement n'est possible en position
5, où toutes les substitutions de nucléotides abolissent
complétement la synthèse d'ARN7. En revanche, dans les
postion 1, 2 et 6, certain changements semblent être bien toléré.
Pris ensemble, ces résultats suggère que d'autre facteurs,
autres que l'appariement Leader-body, jouent un rôle dans la synthèse
des ARNsgm et que la séquence primaire (ou la structure secondaire)
des TRS pourrait obliger à des préférences de position
de base.
F - La transcription chez les
coronavirus suit un mécanisme à hautes fréqeunces
de recombinaison d'ARN similaire et assisté:
Si le terme de recombinaison est appliqué à l'extension-discontinue
durant la synthèse du modèle de transcritpion du brin
négatif, la partie donneuse serait la partie du coprs du génome
(body), l'accepteur serait la partie Leader de l'ARN génomique
et la brin naissant serait le brin négatif synthétisé
en discontinu. Nagy et Simon (Nagy & Simon, 1997) ont défini
un méchanisme dans lequel le transfert de brin est déterminé
par à la fois la séquence similarité des séquences
entre les ARN parents et des déterminants ARN additionnels, présent
seulement sur un des ARN parentaux. Les résultats des travaux
de Pasternak (Pasternak et al, 2001) suggèrent que la synthèse
discontinue des ARNsgm chez les nidovirus peut être considérée
comme un cas spéciale de haute fréquence de recombinaison
asité par des ARN similaires. Alors que la seule focntion de
la TRS du Leader est d'assurer la fidélité du transfert
de brin par appariement des bases avec la partie 3' du brin naissant,
la TRS du body dans l'echantillon donneur possède en plus des
fonctions séquences-spécifiques. Une de ces fonctions
serait de mettre en pause (ou de terminer) la synthèse du brin
naissant et fournir l'opportunité du transfert de brin. De plus,
les nucléotides de la TRS du body pourraient jouer un rôle
dans la réinitiation de la synthèse du brin naissant sur
l'echantillon accepteur.
G - La protéine en doigt
de zinc nsp1 est lié à la transcription:
L'expression du génome des virus à ARN+ commence par la
traduction plutôt que par la transcription. Pour certain virus,
le génome est le seul ARNm viral et l'expression est régulée
au niveau traductionnel et par une protéolyse limitée
des polyprotéines. D'autres virus, comme comme nous avons vu
chez les nidovirus, génère également des ARNm subgénomiques.
La transcription des nidovirus n'est pas essentielle pour la réplication
du génome. Il a été montré, par Tijms et
son équipe (Tijms et al., 2001), que la sous-unité N-terminal
de la réplicase, la protéine 1 non structurale (nsp1)
du nidovirus EAV n'est pas indispensable pour la réplication
mais cruciale pour la transcription, en couplant l'expression de la
réplicase et la synthèse des ARNsgm. Nsp1 est composée
de deux domaines à protéase papaïne-like (PCPa
et PCPb) et d'un doigt de zinc N-terminal
prédit, lequel était impliqué dans la transcription
par mutagénèse dirigée.L'intégrité
structural de nsp1 est essentielle, suggérant que les domaines
protéases forme une plateforme pour le doigt de zinc afin qu'il
opère dans la transcription.
Le premier produit de la réplication "multiproccessing"
de l'EAV est nsp1, une protéine de 260 a.a.qui se libère
elle-même co-traductionnellement par une PCPb
en sa moitié C-terminale. Pour étudier le rôle de
nsp1durant le cycle de vie de l'EAV, un mutant de l'ANDc infectieux
de l'EAV knock-out pour nsp1 a été construit.(Marieke
et al; 2001). Deux mutants ont été construits:

Figure 7 : Construction des mutants "knock-out" Dnsp1
du virus EAV Arterivirus Equine Arteritis
(d'après Tijms, M. A. et al. 2001)
Chez l'un (D0310), une supression
des nucléotides 297 à 1004 aboutit à un arrêt
de synthèse des ARNsgm, mais n'affecte pas la réplication
du génome. Chez le second mutant (Dnsp1),
l'expression de nsp1 a été incativée sans modifier
la séquence en 3' du Leader (stem loop). Pour ceci, le codon
d'initiation de la traduction (les nucléotides 225 à 227)
de l'ORF1a ont été remplacé par AUGAinsi, nsp1
n'est plus traduite et la réplicase démarrer avec nsp2
à partir du codon d'un AUG construit en amont de la séquence
codant pour nsp2. Les résultats (cf figure 8)
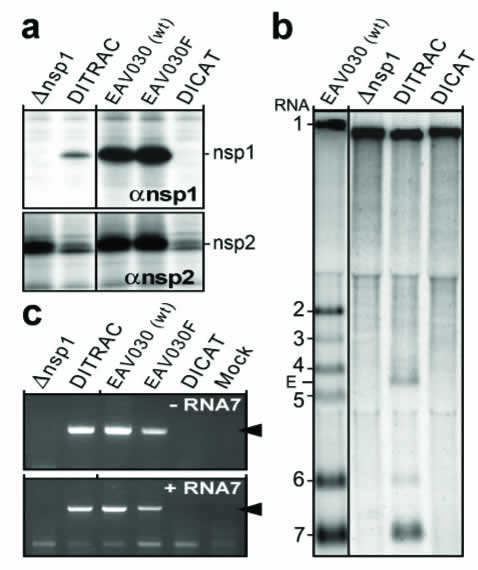
Figure 8 : nsp1 est un facteur essentiel pour la transcription mais
non pour la réplication du génome de l'EAV (d'après
Tijms, M. A. et al. 2001)
indiquent que la protéine nsp1, les ARNm (comme
l'ARN7) et les ARNsgm sont produit uniquement lorsque celà est
nécessaire.
Pour mieu déterminer le rôle de nsp1, la protéine
a été produite en utilisant une cassette d'expression
sous contrôle d'un IRES (Internal Ribosome Entry Site) introduit
en fin de l'ORF21b (figure 9).

Figure 9 : Trans-complementation de la fonction de nsp1 pour la
transcription
(d'après Tijms, M. A. et al. 2001)
Une trans-complémentation de la fonction de
nsp1 pour la transcription a été observée.
Une comparaison assitée par ordinateur des séquences de
la protéine nsp1 d'artérivirus ont révélée
la présence d'un motif en doigt de zinc (Zinc Finger ou ZF) ,
et les 2 domaines protéase (PCPa et
PCPb) (figure 10) dans la séquence
de nsp1.

Figure 10 : Identification d'un motif en doigt de zinc dans le
domaine N-terminal de la réplicase des arterivirus (d'après
Tijms, M. A. et al. 2001)
Les motif ZF sont ubiquitaires dans les facteurs de
transcription. Une analyse par mutation-délétion de ces
domaines a amené à conclure que le motif ZN est utilisé
lors de la transcription discontinue. En réalité, une
mutation des résidus Cys/His (residus typique des motifs en doigt
de zinc) mène à une transcription négative chez
ces mutants seulement lorsque l'activité des domaines ZF été
détruite.
H - La réplication de
l'ARN viral:
Contrairement à la synthèse discontinue de l'ARNm, la
production de l'ARN génomique (brin entier positif) requiert
une synthèse sans interruption en utilisant des matrices (template)
négatives. Ainsi, le mécanisme de réplication de
l'ARN et la transcription de l'ARNm subgénomique diffère
à certain égard. L'étude de la réplication
de l'ARN a été facilitée par DI RNA cloné,
lequel peut répliquer à l'aide de la machinerie de réplication
fournie par un virus MHV sauvage. La réplication de DI RNA a
été utilisée pour déterminer des séquences
d'approximativement 400 nucléotides aux extrémités
3' et 5' final de l'ARN génomique. Durant la réplication,
la séquence Leader du virus peut être rapidement remplacé
par celle du virus helper, suggérant que la réplication
de l'ARN implique également une matrice d'ARN libre de manière
similaire à la transcription. Cependant, le signal cis-déclenchant
(cis-activating) pour la réplication est différent de
celui de la transcription. Le mécanisme précis de la réplication
de coronavirus reste encore à déterminer.
I - La
traduction des protéines virales :
Bien que tous sauf le plus petit des ARNm du coronavirus soient polycistroniques,
en général, seul l'ORF en 5 ' de chaque ARNm est traduite.Récemment,
quelques exception à cette généralisation ont été
découvertes. Comme décris ci-dessus, chez tous les coronavirus,
l'ARNm1 contient 2 grandes ORF qui chevauche de 43 à 76 nucléotides
et cet ARNm est traduit en une seule polyprotéine par un mécanime
de décalage du cadre de lecture (frameshifting). La polyprotéine
subirait des modifications (process) co- et post-traductionelle par
des protéases virales et de l'hôte amenant à de
multiples protéines. Plusieurs autres ARNm des coronavirus sont
dicistroniques ou tricistroniques. Par exemple, l'ARN3 de l'IBV (avian
coronavirus Infectious Bronchitis Virus) contient trois ORFs légèrement
chevauchantes qui sont toutes les trois traduites in vivo et
in vitro. La traduction de ces trois ORF est régulée
par un IRES en amont qui permet aux ribosomes de dévier (bypass)
en amont des ORF et de traduire l'ORF3C par un mécanisme de traduction
cap-indépendant similaire à la traduction chez les Picornavirus.
La protéine légèrement hydrophobe de 10-12 kd codée
par l'ORF3C de l'IBV pourrait être une protéine de l'enveloppe
virale appelé E, similaire à la protéine TGEV codée
par l'ARNm4 monocistronique et par le second ORF de l'ARNm5 du MHV.
L'ARNm5 de l'IBV contient deux ORFqui sont toutes les deux traduites
in vitro et in vivo, et une ORF interne avec laquelle
le gène N du BCoV (Bovine CoronaVirus) est traduit in vivo.
Le nombre d'ORF codant pour des protéines non-structurales, leurs
ordres dans le génome, et leurs mécanismes de traduction
diffèrent en proportion des espèces de coronavirus. Les
fonctions des protéines non-structurales sont largement inconnues.
Plusieurs protéines non-structurales, comme par exemple les gènes
2,4 et 5a du MHV, ne sont pas essentielles pour la production du virus
in vitro.
VI- LES CORONAVIRUS, CAUSES DU SYNDROME RESPIRATOIRE
AIGU SEVERE (SRAS ou SARS pour SEVERE ACUTE RESPIRATORY SYNDROME)
Ce qui est connu à l'heure actuelle:
Le SARS est un typede virus penumonique, dont les symptômes
inclus la fièvre, une toux sèche, la dyspnée (détresse
respiratoire), le mal de tête, et l'hypoxémie (faible concentration
en oxygène dans le sang). Les résultats typiques des laboratoires
incluent également la lymphopénie (réduction du
nombre de lymphocytes) et des niveaux modérément élevés
d'aminotransférase (indiquant des dommages au niveau du foie).
La mort pourrait résulter de la perte progressive de respiration
due à un endomagement des alvéoles. Le cours clinique
typique de SARS comporte une amélioration des symptômes
pendant la première semaine de l'infection, suivie d'une détérioration
pendant la deuxième semaine. Les études indiquent que
cette détèrioration pourrait être liée aux
immuno-réactions plutôt qu'à la réplication
virale non contrôlée chez le patient
Microspcopie electronique du virus SRAS
(d'après le
département de Microbiology & Immunology de l'université
de Leicester)
Le virus s'est manifesté en février 2003
dans la province de Guangdong en Chine, où 300 personnes sont
tombées malades, et au moins cinq en sont mortes. Après
des premiers signes tendant à montrer qu'un paramyxovirus serait
responsable, la cause réèle à semblait être
un nouveau coronavirus aux propriétés peu communes. D'un
coté, le virus du SARS peut posser en cellules Vero (une lignée
de cellules de fibroblaste isolée en 1962 chez un primate) -
une propriété nouvelle pour les HCoV, lesquels ne peuvent
pas être cultivés. L'infection par ce virus sur ces cellules
déclenche un effet cytopathe, et le bourgeonnement de coronavirus-comme
des particules du réticulum endoplasmique dans les cellules infectées.
Le virus de SARS est connu pour être transmis
via des gouttelettes produites par la toux et l'éternuement,
mais d'autres voie d''infection peuvent également être
impliqués, comme la contamination fécale : dans l'article
de Donnelly
CA, et al. Epidemiological determinants of spread of causal agent of
severe acute respiratory syndrome in Hong Kong. Lancet volume 361, 03
May 2003, les auteurs rapportent que:
- Les symptomes rapportés les plus commun sont la fièvre
(94%), avec entre 51 et 72% des patients présentant des symptômes
similaires à la grippe, froids, malaise, perte d'appétit,
et myalgie (endolorissement d'un muscle, ou d'un groupe musculaire).
Les symptômes gastro-intestinaux sont détecté
mais moins souvent, y compris la diarrhée (27%), le vomissement
(14%), et la douleur abdominale (13%).
- La durée moyenne d'incubation du SRAS serait comprise
entre 4 à 6 jours.
- Le taux de cas fatale pour les personnes de moin de 60 ans serait
de 2 à 13%, tandis qu'il serait compris entre 3 et 3% pour
les plus de 60ans, avec en moyenne un taux de mortalité de
5%.
- Les faisceaux de cas ont joué un rôle important au
cours de l'épidémie
 L'amplification
de petites régions du gène de la polymérase ( la
partie la plus conservée du génome des coronavirus) ,
par RT-PCR (Reverse Transcriptase Polymerase Chain Reaction) et séquençage
des nucléotides ont révélé que le virus
du SRAS est un nouveau coronavirus qui n'avait encore jamais était
détecté dans la population humaine. Cette conclusion a
été confirmé par des tests sérologiques.
Nous connaissons maintenant la séquence
complète (environ 29.700 nucléotides) de plusieurs
isolats du virus du SARS. La séquences semble être typique
des coronavirus, hors évidemment des structures non-habituelle,
bien qu'il y ait quelques différences dans la composition des
protéines non-structurales qui sont inhabituelle.

Organisation du Coronavirus associé au Syndrome Respiratoire
Aigue Sévère (SRAS ou SARS pour Severe Acute Respiratory
Syndrome), (d'après Paul A. Rota et al, Science. 2003 May 1)
Il n'y a pour l'instant aucun consensus sur le fait
que des drogues antivirales se soient avérées efficaces
dans le traitement contre le SARS ou contre n'importe quelle infection
par un coronavirus. Aucun vaccin n'a encore été développé
contre le SARS. Cependant, de nouvelles drogues visant spécifiquement
ce virus sont en cours de développement. Une cible de ces drogues
pourrait être la Proteinase (3CLpro) dont la structure
a récemment été déterminée:
|
|
| 2.54 Å: structure
crytallisée de la proteine du HCoV 229E Mpro
|
HCoV Mpro forme un dimère serré (interface
de contact, principalement entre le domaine II de la molécule
A et des résidus de NH2-terminal de la molécule B
: ~1300 Å) dans le cristal |
(d'après Kanchan Anand et al., Science 13 May 2003)
 La
réalisation de la structure tridimensionnele de cette protéine
a permis de se pencher sur l'élaboration d'inhibiteur du site
catalytique de cette protéase: un composé appelé
AG7088 (ou p-fluoro-benzyl) serait éventuellement un bon candidat
comme inhibiteur de cette protéinase. La
réalisation de la structure tridimensionnele de cette protéine
a permis de se pencher sur l'élaboration d'inhibiteur du site
catalytique de cette protéase: un composé appelé
AG7088 (ou p-fluoro-benzyl) serait éventuellement un bon candidat
comme inhibiteur de cette protéinase.
La superposition des deux complexes suggère
que les chaînes latérales d'AG7088 se liant aux sous-site
S1 et S4 pourrait être adapté pour inhiber la protéine
Mpro des coronavirus.
(d'après Kanchan Anand et al., Science 13 May 2003)
Les tests diagnostiques pour les infections par des
coronavirus sont de deux types:
- Des tests sérologiques sur les anticorps anti-coronavirus
par ELISA (Enzyme-Linked Immunosorbent Assays) détectant
les anticorps produit en réponse à l'infection. Bien
que certain patient n'est pas d'anticorps anti-coronavirus détectables
14 jours après le début de la maladie, l'interprétation
définitive des essais négatifs d'anticorps de coronavirus
n'est possible que pour les spécimens obtenus au delà
de 21 jours après le début de la fièvre.
- Les tests moléculaires consitent en des tests de RT-PCR
specifique pour l'ARN de ce nouveau Coronavirus. Ils peuvent détecter
l'infection dans les 10 jours après le début de la
fièvre chez les patients atteint de certain SARS, mais le
temps de la diminution de la virémie et du virus reste inconnue.
Aussi, des essais de RT-PCR réalisés trop tard pourrait
donner des résultats négatifs. Des tests de diagnostiques
commerciaux sont maintenant disponibles.
Enfin récemment, des chercheurs ont trouvé
des anticorps du virus provoquant le syndrome respiratoire aigu sévère
(SRAS) chez cinq professionnels spécialisés dans le commerce
des animaux sauvages, mais aucun d'entre eux n'aurait développé
de symptômes.
Cette découverte semblerait montrer que la forme du coronavirus
qu'on soupçonne avoir franchi la barrière des espèces,
soit depuis la civette, soit depuis le raton laveur, vers l'homme, serait
moins dangereuse que le coronavirus du SRAS que se transmettent les
humains entre eux et qui a fait près de 700 morts parmi les 8.000
contaminés dans le monde.
Après être passé par l'animal, le virus du SRAS
subirait des modifications en pénétrant dans le corps
humain qui augmentent sa dangerosité, expliquent les chercheurs.
Liens sur le SARS:
BIBLIOGRAPHIE:
* Almazan, F., Gonzalez, J. M., Pénzes, Z., Izeta, A.,
Calvo, E., Plana-Duran, J. & Enjuanes, L. (2000). Engineering
the largest RNA virus genome as an infectious bacterial artificial chromosome.
Proc. Natl. Acad. Sci. USA 97, 5516-5521.
* Alonso, S., Izeta, A., Sola, L & Enjuanes, L. (2002a).
Transcription regulatory sequences and mRNA expression levels in the
coronavirus transmissible gastroenteritis virus. J. Viral. 76, 1293-1308.
Alonso, S., Sola, 1., Teifke, J., Reimann, 1., Izeta, A., Balach,
M., Plana- Duran, J., Moormann, R. J. M. & Enjuanes, L. (2002b).
In vitro and in vivo expression of foreign genes by transmissible gastroenteritis
coronavirus-derived minigenomes. J. Gen. Viral. 83, 000-000.
Castilla, J., Pintado, B., Sola, 1., Sanchez-Morgado, J. M.
& Enjuanes, L. (1998). Engineering passive irnmunity in
transgenic mice secreting virus-neutralizing antibodies in mi1k. Nature
Biotech. 16, 349-354.
Delmas, B., Gelfi, J., L'Haridon, R., Vogel, L. K., Norén,
O. & Laude, H. (1992). Aminopeptidase N is a major receptor
for the enteropathogenic coronavirus TGEV. Nature 357, 417-420.
Den Boon, J. A., Kleijnen, M. F., Spaan, W. J. M. & Snijder,
E. J. (1996).
Equine arteritis virus subgenomic rnRNA synthesis: analysis of leader-body
junctions and replicative-fonn RNAs. J. Viral. 70,4291-4298.
Enjuanes, L., Brian, D., Cavanagh, D., Holmes, K., Lai, M. M.
C., Laude, H., Masters, P., Rottier, P., Siddell, S. G., Spaan, W. J.
M., Taguchi, F. & Talbot, P. (2000a). Coronaviridae. In
Virus taxonomy. Classification and nomenclature of viruses, pp. 835-849.
Edited by M. H. V. van Regenmortel, C. M. Fauquet, D. H. L. Bishop,
E. B. Carsten, M. K. Estes, S. M. Lemon, D. J. McGeoch, J. Maniloff,
M. A. Mayo, C. R. Pringle & R. B. Wickner. New York: Academic Press.
Enjuanes, L., Sola, 1., Almazan, F., Ortego, J., Izeta, A.,
Gonzalez, J. M., Alonso, S., Sanchez-Morgado, J. M., Escors, D., Calvo,
E., Riquelme, C. & Sanchez, C. M. (2001). Coronavirus derived
expression systems. Journal of Biotechnology 88, 183-204.
Enjuanes, L., Spaan, W., Snijder, E. & Cavanagh, D. (2000b).
Nidovirales. In Virus taxonomy. Classification and nomenclature ofviruses,
pp. 827-834. Edited by M. H. V. van Regenmortel, C. M. Fauquet, D. H.
L. Bishop, E. B. Carsten, M. K. Estes, S. M. Lemon, D. J. McGeoch, J.
Mani1off, M. A. Mayo, C. R. Pringle & R. B. Wickner. New York: Academic
Press.
Escors, D., Ortego, J., Laude, H. & Enjuanes, L. (2001a).
The membrane M protein carboxy terminus binds to transmissible gastroenteritis
coronavirus core and contributes to core stabi1ity. J. Viral. 75, 1312-1324.
Escors, D., Ortego, J., Laude, H. & Enjuanes, L. (2001b).
Organization of two transmissible gastroenteritis coronavirus membrane
protein topologies within the virion and core. J. Viral. 75, 12228-12240.
Gonzalez, J. M., Penzes, Z., Almazan, F., Calvo, E. & Enjuanes,
L. (2002). Stabi1ization of a full-length infectious cDNA clone
of transmissible gastroenteritis coronavirus by the insertion of an
intron. J. Viral. In press.
Gorbalenya, A. E. (2001). Big nidovirus genome. When
count and order of domains matter. In The Nidoviruses (Coronaviruses
and Arteriviruses), pp. 1-17. Edited by E. Lavi, S. Weiss & S. T.
Hingley. New York: Kluwer Academic/Plenum Publishers.
Holmes, K. V. & Lai, M. M. C. (1996). Coronaviridae:
the viroses and their replication. In Fundamental Virology, 3rd edn,
pp. 541-559. Edited by B. N. Fields, D. M. Knipe & P. M. Howley.
Philadelphia: Lippincott-Raven, PA.
Izeta, A., Smerdou, C., Alonso, S., Penzes, Z., Méndez,
A., Plana-Duran, J. & Enjuanes, L. (1999). Replication
and packaging of transmissible gastroenteritis coronavirus-derived synthetic
minigenomes. J. Virol. 73, 1535-1545.
Lai, M. M. C. & Cavanagh, D. (1997). The molecular
biology of coronaviruses. Adv. Virus Res. 48, 1-100.
Masters, P. S. (1999). Reverse genetics of the largest
RNA viroses. Adv. Virus Res. 53, 245-264.
Nagy, P. D. & Simon, A. E. (1997). New insightsinto
the mechanisms of RNA recombination. Virology 235, 1-9.
Pasternak, A. O., Gultyaev, A. P., Spaan, W. J. & Snijder,
E. J. (2000). Genetic manipulation of arterivirus alternative
rnRNA leader-body junction sites reveals tight regulation of structural
protein expression. J. Virol. 74, 11642-11653.
* Pasternak, A. O., van den Born, E., Spaan, W. J. M. &
Snijder, E. J. (2001). Sequence requirements for RNA strand
transfer during nidovirus discontinuous subgenomic RNA synthesis. EMBO
J. 20, 7220-7228.
Sanchez, C. M., Izeta, A., Sanchez-Morgado, J. M., Alonso, S.,
Sola, 1., Balasch, M., Plana-Duran, J. & Enjuanes, L. (1999).
Targeted recombination demonstrates that the spike gene of transmissible
gastroenteritis coronavirus is a determinant of its enteric tropism
and virulence. J. Virol. 73,7607-7618.
Sawicki, D. L., Wang, T. & Sawicki, S. G. (2001).
The RNA structures engaged in replication and transcription of the A59
strain of mouse hepatitis virus. J. Gen. Virol. 82, 386-396. .
Sawicki, S. G. & Sawicki, D. L. (1990). Coronavirus
transcription: subgenomic mouse hepatitis virus replicative intermediates
function in RNA synthesis. J. Virol. 64, 1050-1056.
Sola, 1., Castilla, J., Pintado, B., Sanchez- Morgado, J. M.,
Whitelaw, B., Clark, J. & Enjuanes, L. (1998). Transgenic
mice secreting coronavirus neutralizing antibodies into the milk. J.
Virol. 72, 3762-3772.
* Tijms, M. A., van Dinten, L. C., Gorbalenya, A. E. & Snijder,
E. J. (2001). A zinc finger-containing papain-like protease
couples subgenomic rnRNA synthesis to genome translation in a positive-stranded
RNA virus. Proc. Natl. Acad. Sci. USA 98, 1889-1894.
* van Marie, G., Dobbe, J. C., Gultyaev, A. P., Luytjes, W.,
Spaan, W. J. M. &
Snijder, E. J. (1999). Arterivirus discontinuous rnRNA transcription
is guided by base pairing between sense and antisense transcription-regulating
sequences. Proc. Nat. Acad. Sc. USA 96, 12056-12061.
|
 L'amplification
de petites régions du gène de la polymérase ( la
partie la plus conservée du génome des coronavirus) ,
par RT-PCR (Reverse Transcriptase Polymerase Chain Reaction) et séquençage
des nucléotides ont révélé que le virus
du SRAS est un nouveau coronavirus qui n'avait encore jamais était
détecté dans la population humaine. Cette conclusion a
été confirmé par des tests sérologiques.
Nous connaissons maintenant la séquence
complète (environ 29.700 nucléotides) de plusieurs
isolats du virus du SARS. La séquences semble être typique
des coronavirus, hors évidemment des structures non-habituelle,
bien qu'il y ait quelques différences dans la composition des
protéines non-structurales qui sont inhabituelle.
L'amplification
de petites régions du gène de la polymérase ( la
partie la plus conservée du génome des coronavirus) ,
par RT-PCR (Reverse Transcriptase Polymerase Chain Reaction) et séquençage
des nucléotides ont révélé que le virus
du SRAS est un nouveau coronavirus qui n'avait encore jamais était
détecté dans la population humaine. Cette conclusion a
été confirmé par des tests sérologiques.
Nous connaissons maintenant la séquence
complète (environ 29.700 nucléotides) de plusieurs
isolats du virus du SARS. La séquences semble être typique
des coronavirus, hors évidemment des structures non-habituelle,
bien qu'il y ait quelques différences dans la composition des
protéines non-structurales qui sont inhabituelle.














 La
réalisation de la structure tridimensionnele de cette protéine
a permis de se pencher sur l'élaboration d'inhibiteur du site
catalytique de cette protéase: un composé appelé
AG7088 (ou p-fluoro-benzyl) serait éventuellement un bon candidat
comme inhibiteur de cette protéinase.
La
réalisation de la structure tridimensionnele de cette protéine
a permis de se pencher sur l'élaboration d'inhibiteur du site
catalytique de cette protéase: un composé appelé
AG7088 (ou p-fluoro-benzyl) serait éventuellement un bon candidat
comme inhibiteur de cette protéinase.